La Société Française d’Ecologie et d’Evolution (SFE2) vous propose ce regard de Vincent Miele, Stéphane Dray et Olivier Gimenez, respectivement ingénieur et directeurs de recherche au CNRS, sur l’utilisation de l’Intelligence Artificielle et du ‘deep learning’ en écologie.
MERCI DE PARTICIPER à ces regards et débats sur la biodiversité en postant vos commentaires et questions sur les forums de discussion qui suivent les articles; les auteurs vous répondront.
Images, écologie et deep learning
Vincent Miele (1), Stéphane Dray (2) et Olivier Gimenez (3)
(1) : Ingénieur de Recherche CNRS, Laboratoire de Biométrie et Biologie Evolutive (LBBE), Lyon
(2) : Directeur de Recherche CNRS au sein du LBBE
(3) : Directeur de Recherche CNRS au sein du Centre d’Ecologie Fonctionnelle et Evolutive (CEFE), Montpellier
Regard R95, édité par François Massol
——-
Mots clés : Apprentissage profond, méthodes, deep learning, données massives, images et vidéos, intelligence artificielle, piégeage photographique, reconnaissance d’espèces, vision par ordinateur
——–
- Les images à la croisée des chemins
- Problématiques de vision par ordinateur pour l’écologie
- Intelligence artificielle, vision par ordinateur, apprentissage automatique et deep learning
- Quand les problèmes arrivent
- Conclusions
- Glossaire
- Remerciements
- Bibliographie
- Pour aller plus loin, regards connexes
- Forum de discussion sur ces deux regards
——
Les images à la croisée des chemins
L’écologie est témoin d’une excitante convergence. D’un côté, la communauté produit massivement des données de type image, depuis les pièges photos/vidéos, les images aériennes de drones ou de satellites, les données LIDAR (mesures de distance par faisceau lumineux), les photos/vidéos sous-marines jusqu’aux images de laboratoire. Ces données proviennent de campagnes d’acquisition de données sur tout le spectre de l’écologie et des milieux/organismes. De l’autre côté en intelligence artificielle, une nouvelle génération de modèles mathématiques et informatiques a bouleversé la vision par ordinateur, c’est-à-dire l’automatisation de la compréhension du contenu d’une image par la machine.
Ces approches dites d’apprentissage profond* ou deep learning* (terme que nous utiliserons par la suite ; Baraniuk, Donoho et Gavish, 2020) ont révolutionné le domaine depuis une fameuse compétition en reconnaissance d’images remportée en 2012 par Alex Krizhevsky (Krizhevsky, Sutskever et Hinton, 2012). Leur principe est simple : construire un modèle qui synthétise un jeu de données dit d’entraînement préalablement analysé à la main – on dira que les images sont annotées – de sorte que la machine pourra utiliser ce modèle pour réaliser par elle-même le traitement de nouvelles données, en masse si besoin. Utilisé dans de nombreuses disciplines scientifiques (par exemple en imagerie médicale) mais aussi extrêmement « tendance » dans les médias, le deep learning* est un outil prometteur pour le traitement automatisé d’images en écologie (Christin, Hervet et Lecomte, 2019 ; Lamba et al., 2019).
Dans cet article, nous discuterons de cette excitante synchronie entre données produites et méthodologie à disposition. En particulier, le transfert de cette méthodologie vers notre communauté nécessite la découverte d’une nouvelle terminologie, de nouveaux modèles mathématiques et algorithmes, ainsi que de nouvelles techniques de programmation. Cette somme de connaissances est toutefois rarement décrite d’une façon abordable pour les non-spécialistes. Pour l’écologue, découvrir ces techniques représente un effort certain, l’enjeu étant de savoir si cet effort peut s’avérer rentable. Au sein du groupe d’Ecologie Statistique de la SFE2, nous proposons ici une invitation à évaluer la pertinence de l’utilisation de ces méthodes et outils dans notre domaine.
Problématiques de vision par ordinateur pour l’écologie
Plusieurs problématiques d’analyse d’images en écologie reviennent à des problèmes d’apprentissage classiques en vision. Dans la majorité des cas, il s’agit d’entraîner l’ordinateur pour qu’il puisse réaliser une tâche répétitive mais faisable par l’humain, par exemple identifier une espèce de mésange sur une photo. On vise ici des gains de temps pour passer à l’échelle de centaines de milliers d’images, pour éviter la pénibilité du traitement manuel ou proposer un complément aux approches de sciences citoyennes (production participative) pour l’annotation d’images (Torney et al., 2019).
L’essor des pièges-photos a conduit aux premières applications du deep learning* en écologie. Il s’agit le plus souvent d’identification spécifique, afin d’identifier l’espèce d’un individu présent (en entier ou partiellement) sur une photo, en supposant ici qu’il n’y ait qu’une espèce. Ceci revient à un problème classique de classification* où l’algorithme doit prédire la classe de chaque image analysée (ici, l’espèce associée à chacune) avec une valeur de confiance (cf. Figure 1). Les écologues, naturalistes ou citoyens annotent un ensemble d’images le plus grand possible, en précisant quelle espèce est présente sur quelle image, puis les algorithmes prennent le relais pour apprendre par eux-mêmes une règle de classification utilisable sur de nouvelles images. Nous considérerons par la suite que nous avons en main des centaines d’images de fleurs qui correspondent à trois clades –la pâquerette, l’iris et la tulipe– et que nous voulons mettre au point un programme informatique qui prendra en entrée une image et retournera automatiquement le nom de la fleur trouvée. Cette approche a été largement mise en place, depuis des études séminales sur les grands mammifères de savane (Norouzzadeh et al., 2018 ; Willi et al., 2019) jusqu’à l’étude de différentes espèces d’insectes (Ärje et al., 2020) ou de poissons (Villon et al., 2018). Le projet Pl@ntNet est par ailleurs le meilleur exemple de système d’identification dédié aux plantes (près de 30000 espèces).
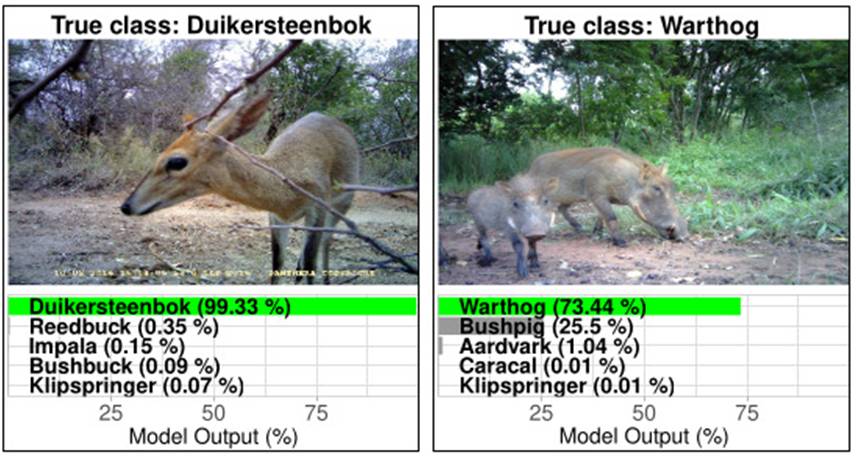
Figure 1 : Identification spécifique sur images de piège-photo.
(Source : Willi et al., 2019 ; image reproduite avec autorisation de l’auteur.)
Toutefois, une image peut être hétérogène, c’est-à-dire contenir plusieurs éléments, possiblement de plusieurs classes d’intérêt (par exemple plusieurs espèces). Les algorithmes de détection d’objet sont alors régulièrement utilisés. Leur principe est de détecter l’élément et de l’encadrer d’un rectangle (Figures 2 et 3), comme par exemple chaque animal présent dans une image de piège photo (Schneider, Taylor et Kremer, 2018 ; Beery, Morris et Yang, 2019 ; logiciel MegaDetector). Dans notre exemple, cette approche s’avérerait nécessaire si une image pouvait contenir à la fois des iris et des tulipes qu’il faudrait déceler. Ce principe est également repris pour détecter une sous-partie d’un organisme (on parle alors de recadrage ou cropping), par exemple les flancs de girafes (Miele et al., 2021) ou les visages de chimpanzés (Schofield et al., 2019). A nouveau, l’écologue constitue le jeu de données d’entraînement avec des images pour lesquelles les éléments d’intérêt sont encadrés manuellement avec l’interface graphique d’un logiciel (par exemple VIA, Dutta et Zisserman, 2019).

Figure 2 : Comptage de baleines à partir d’images aériennes.
(Adapté de Guirado et al., 2019, CC BY 4.0.)
La même idée s’applique quand il s’agit de réaliser un comptage à partir d’une image. Cette approche a été mise en œuvre dans différents contextes, pour compter des gnous (Torney et al., 2019) ou des baleines (Guirado et al., 2019 ; Figure 2) dans des images aériennes ou satellites, des oursins dans des images sous-marines (Huang et al. 2019), mais aussi tout autre élément énumérable compte tenu de la résolution de l’image (par exemple des graines par Masteling et al., 2020 ; Figure 3). A noter que des difficultés supplémentaires se présentent quand les éléments à compter ne sont visibles que sur quelques pixels (Duporge et al., 2020).
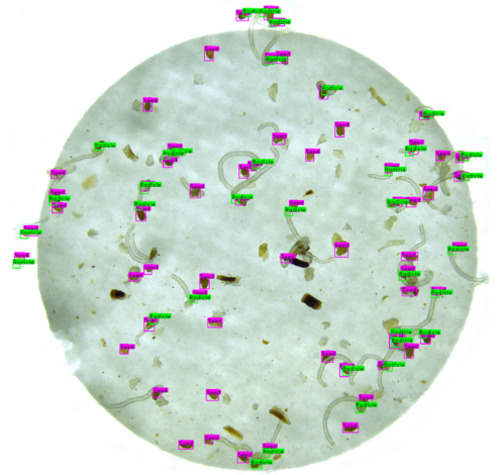
Figure 3 : Comptage de graines dans un gel.
(Adapté de Masteling et al., 2020, CC BY 4.0.)
Sur des images hétérogènes, il peut aussi s’agir de délimiter différentes zones dans l’image, comme différents types de sol, de végétation ou d’habitats (Figure 4 ; Kattenborn, Eichel et Fassnacht, 2019). Le problème revient à de la segmentation d’image où chaque pixel est classé par l’algorithme d’apprentissage dans une des classes d’intérêt (par exemple corail/sable/eau dans Brodrick, Davies et Asner, 2019 ou végétal/non non-végétal dans Abrams et al., 2019). Bien sûr, sans ce cadre, l’effort d’annotation des données d’entraînement par l’écologue s’avère très conséquent puisque chaque image doit être segmentée manuellement.

Figure 4 : Segmentation de végétation.
(Adapté de Kattenborn et al., 2019, CC BY 4.0.)
Par ailleurs, la détection de points d’intérêt dans une image peut permettre d’étudier le profil morphologique d’un individu. Par exemple, l’algorithme peut apprendre à prédire les points de jonction entre les membres d’un animal. Ainsi, de récentes approches ont permis d’estimer la position des différentes parties du corps d’une souris ou d’une drosophile (Mathis et al., 2018 ; Pereira et al., 2019 ; Graving et al., 2019). On parle alors d’estimation de pose. Ces approches ouvrent des perspectives pour l’étude à grande échelle du mouvement corporel et du comportement animal.
Le deep learning* permet également d’envisager la reconnaissance individuelle animale par image (Schneider et al., 2019 ; Figure 5). Traditionnellement, cette reconnaissance s’est appuyée sur la recherche de caractéristiques (features) comme la forme des taches dans le pelage des girafes (Bolger et al., 2012). Des travaux récents ont emprunté des algorithmes de reconnaissance faciale pour les adapter au traitement du visage animal (par exemple mandrill, Charpentier et al., 2020) : ces algorithmes, appliqués à la reconnaissance individuelle humaine, soulèvent toutefois des questions d’éthique sur le respect de la vie privée. D’autres études ont reformulé la problématique de la classification à une problématique de reconnaissance individuelle : les classes d’intérêt sont les différents individus, par exemple des individus baleines (Bogucki et al., 2019), girafes (Miele et al., 2021) ou oiseaux (Ferreira et al., 2020). On notera toutefois que ces méthodes se heurtent au problème de l’identification d’individus non connus a priori (c’est-à-dire non présents dans le jeu d’entraînement), nécessaire à la mise en place concrète d’une approche par capture-recapture photographique.

Figure 5 : Reconnaissance individuelle de chimpanzés.
(Adapté de Schofield et al., 2019, CC BY 4.0.)
Bien que les techniques de deep learning* soient majoritairement dédiées aux images, les sons peuvent également être analysés par ces techniques, en particulier en écologie. Le son peut être directement traité par un modèle spécifique préalablement entraîné sur une immense banque de sons (par exemple l’ensemble de YouTube) pour classifier des ambiances sonores de différents écosystèmes (Sethi et al., 2020). Mais plusieurs études ont proposé de travailler sur les sons vus comme des images dans une représentation temps/fréquence/intensité (un spectrogramme*). Différents sons induisent différentes images, et il est alors possible de revenir à une problématique de classification d’images pour permettre la détection de signaux spécifiques (par exemple présence d’oiseaux dans Stowell et al., 2019 ou écholocalisation de chauves-souris dans Mac Aodha et al., 2018 ; Figure 6).

Figure 6 : Détection de signaux dans le son
(Adapté de Mac Aodha et al., 2018, CC BY 4.0.)
Intelligence artificielle, vision par ordinateur, apprentissage automatique et deep learning
Les différentes applications de la reconnaissance d’images en écologie ont bénéficié de la révolution qui s’est opérée récemment dans le domaine du deep learning*. De quoi s’agit-il ? Pour le comprendre, il nous faut d’abord évoquer les finesses sémantiques de ce domaine, depuis l’intelligence artificielle vers l’apprentissage automatique* (machine learning) et profond* (Figure 7). Bien avant de devenir un « buzzword », l’intelligence artificielle est d’abord un champ disciplinaire scientifique bien identifié (Konieczny et Prade, 2020), qui s’intéresse aux techniques permettant à un ordinateur/système d’effectuer une tâche ou résoudre un problème qui nécessite usuellement l’intelligence humaine.
On retrouve des sous-disciplines comme la robotique et le traitement automatique du langage, ainsi que la vision par ordinateur et l’apprentissage automatique qui nous concernent ici. Ce dernier terme caractérise un ensemble de méthodes relevant de la statistique, des mathématiques, de l’informatique et de l’algorithmique : peut être qualifié d’apprentissage automatique tout algorithme qui a la capacité d’apprendre automatiquement des structures dans les données sans qu’elles soient nécessairement explicitées. Dit plus simplement : on ne dit pas à l’algorithme ce qu’il doit chercher, on le laisse chercher/trouver des structures d’intérêt par lui-même dans les données.
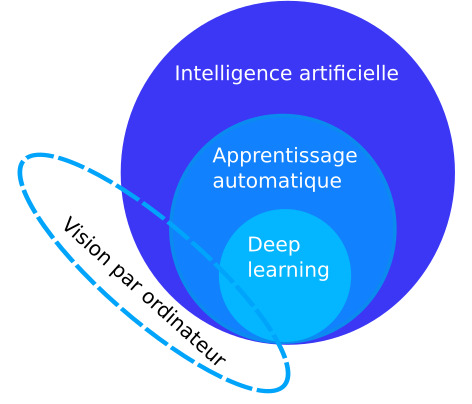
Figure 7 : L’intelligence artificielle est d’abord un champ disciplinaire scientifique qui inclue une partie de la vision par ordinateur ainsi que l’apprentissage automatique* (machine learning). L’apprentissage profond* ou deep learning est un sous-domaine de l’apprentissage automatique. (Schéma V. Miele et al.)
Dans la suite, nous nous restreindrons aux techniques d’apprentissage dit supervisé. Il s’agit d’apprendre à résoudre un problème à partir d’un échantillon de données (le jeu d’entraînement) pour lesquelles la solution est connue, c’est-à-dire apprendre une fonction de prédiction à partir d’exemples annotés. Le modèle linéaire, bien connu des écologues, peut figurer dans cette catégorie. Par exemple, on peut apprendre à reconnaître un iris à partir de la morphologie des fleurs des trois clades étudiés avec un modèle linéaire multinomial* donnant la probabilité de classer une image comme un iris en fonction des angles entre les sépales, du nombre de pétales, etc. Mais revenons à la révolution du deep learning* pour les images. Ce qui est intéressant ici, c’est que de nombreux problèmes de vision par ordinateur vont pouvoir être formulés comme des problèmes d’apprentissage automatique, si bien que deux sous-disciplines de l’intelligence artificielle se rejoignent.
Parmi ces techniques d’apprentissage, les réseaux de neurones sont l’option qui nous intéresse ici. Leur esprit général est d’imaginer qu’en appliquant plusieurs vagues de transformations aux données, l’une après l’autre, on doit pouvoir plonger les données dans un espace mathématique qui facilitera l’apprentissage des règles de prédiction/classification. Dans cette idée, on peut imaginer une vague qui consiste à transformer une image de fleur en un vecteur des fréquences des pixels jaunes, verts et violets. Ensuite il sera aisé d’estimer une règle de prédiction sur ce vecteur pour discriminer les pâquerettes et les iris. Développée dans les années 50, cette approche s’appuie sur le concept de neurone artificiel, une petite boîte qui transforme un vecteur de données en une valeur numérique. Un réseau de neurones sera composé de plusieurs couches de neurones, chaque couche intervenant sur le résultat de la couche précédente (on retrouve l’idée des vagues de transformations). Ceux de la première couche prennent les données brutes en entrée, et ceux de la dernière couche seront les prédictions (des scores dans le cas d’une classification).
Notre modèle linéaire multinomial est un exemple particulier de réseaux de neurones à une couche composée d’un neurone linéaire. Derrière cette architecture compliquée, il s’agit d’imaginer qu’on peut optimiser chaque neurone pour que le modèle arrive à prédire le mieux possible malgré la présence de structures hautement non linéaires dans les données : c’est la phase d’apprentissage.
Une évolution des réseaux de neurones, les réseaux de neurones convolutifs (convolutional neural networks ; CNN*) entrent alors en jeu. Développés dans les années 80, mais ensuite peu utilisés, ils sont repris par Alex Krizhevsky en 2012 lors d’une compétition de reconnaissance d’images qu’il a outrageusement dominé (Krizhevsky, Sutskever et Hinton, 2012). Ces CNNs* sont fondés sur un concept supplémentaire, la convolution, qui consiste à alimenter des neurones non pas avec l’image entière mais avec chaque petit morceau de cette image (par exemple, 9×9 pixels). Deux images de pâquerettes peuvent se trouver globalement très différentes, mais des petits morceaux de ces images vont s’avérer très similaires sur des zones spécifiques comme par exemple les extrémités des pétales.
En pratique, les CNNs* sont composés d’un grand nombre de couches de neurones, si bien que l’on parle alors de réseaux de neurones profonds. Par contraction des divers éléments terminologiques, on parle d’apprentissage profond (deep learning* en anglais). Dans notre exemple, chaque image de fleur sera parcourue par des neurones qui la transformeront en valeurs numériques. Celles-ci seront utilisées pour estimer la meilleure règle de classification qui permettra de prédire le clade présent sur les images pour lesquelles on connaît la solution (c’est-à-dire pâquerette, iris ou tulipe).
Pourquoi cette révolution maintenant ? On peut l’expliquer par une conjonction d’éléments sociétaux et scientifiques :
1/ la disponibilité de masses de données (du fait de l’internet ; en écologie, les initiatives de sciences participatives ont aussi permis de constituer des grosses bases de données comme https://www.zooniverse.org/),
2/ des annotations manuelles réalisées à large échelle du fait d’une organisation du travail libéralisée (la pénibilité de ce travail peut poser des questions, Metz, 2019),
3/ l’amélioration des algorithmes d’apprentissage (par exemple techniques d’augmentation de données),
4/ l’implication des entreprises mastodontes du numérique (Facebook et Google en tête ; voir aussi https://www.microsoft.com/en-us/ai/ai-for-earth),
5/ la diffusion des codes sources ouverts (par exemple via GitHub) et l’émergence d’une immense communauté de développeurs, et enfin
6/ les capacités de calcul parallèle massif sur processeurs graphiques. (Nous y reviendrons.)
Quand les problèmes arrivent
Dans ce qui précède, nous avons esquissé un panorama des applications du deep learning* en écologie, puis nous avons expliqué le principe des méthodes. Dans ce qui suit, nous nous attardons sur les difficultés liées à leur mise en pratique.
La taille et l’exhaustivité du jeu d’entraînement
Pour estimer les paramètres d’un CNN*, il faut des millions d’images annotées. C’est ainsi que les millions de paramètres des modèles les plus connus (ResNet, Inception, MobileNet) ont été estimés. Cette quantité de données est bien sûr hors de portée des écologues. Heureusement, il existe la technique de l’apprentissage par transfert (transfer learning), qui consiste à s’appuyer sur un modèle déjà estimé : en quelque sorte, on adapte un modèle existant à sa question d’intérêt, sans avoir à construire un modèle à partir de zéro (Willi et al., 2019). Il est alors envisageable de modifier les paramètres du modèle existant en réalisant l’apprentissage avec quelques centaines d’images par classe d’intérêt pour des cas très simples (Schneider et al., 2020).
Seulement, ce n’est pas tout : un CNN* synthétise l’information contenue dans le jeu d’entraînement, mais pas au-delà. Plus la variabilité des cas d’utilisation du CNN* est élevée, plus le jeu d’entraînement devra être grand pour inclure cette variabilité au moment de l’apprentissage du modèle. Par exemple, pour prédire qu’une image contient une espèce sur la base de n’importe quelle sous-partie d’un animal, il faut inclure une grande quantité d’images avec des sous-parties de l’animal lors de l’apprentissage. Un autre exemple est la prédiction des images « vides », autrement dit sans les éléments d’intérêts, qui demande un grand nombre d’images d’entraînement associées justement à la configuration « vide » (Willi et al., 2019). Pour notre exemple, un modèle CNN* entraîné uniquement sur les images des trois clades de fleur qui nous intéressent ne saura reconnaître une image sans fleur et prédira l’une des trois espèces.
La variabilité de l’arrière-plan de l’image et des conditions environnementales (nuit/jour, habitats, saisons) peut s’avérer également décisive. Par exemple, une étude montre qu’une vache sur une plage n’est pas reconnue par un modèle qui a été entraîné avec des images de vaches alpines (Beery, Van Horn et Perona, 2018). Le jeu d’entraînement doit aussi bien couvrir les configurations attendues dans les images à traiter ultérieurement, ce qui représente un défi pour les écologues. En résumé, plus il y a de biais dans le jeu d’entraînement (voir Suresh et Guttag, 2019 pour une typologie des risques de biais), plus les techniques d’apprentissage vont produire des erreurs au moment où elles seront utilisées dans des cas concrets sur de nouvelles images (O’Neil, 2016).
Le difficile contrôle des performances du modèle
Il est difficile de s’assurer du pouvoir prédictif et de la généralisabilité (Suresh et Guttag, 2019) d’un CNN*. Dit autrement, il est difficile de savoir à quel point un modèle CNN* sera efficace sur un jeu de données complètement nouveau, par exemple en une nouvelle localisation (Schneider et al., 2020). En effet, les métriques utilisées pour contrôler la performance sont calculées sur des données dites de test qui, généralement, sont acquises dans les mêmes conditions que celles du jeu de données d’entraînement (Wearn, Freeman et Jacoby, 2019). Par ailleurs, les erreurs d’annotations sont possibles, a fortiori puisqu’il est nécessaire de constituer de grands jeux de données (par exemple grâce aux sciences citoyennes, Torney et al., 2019) et, comme pour bien d’autres méthodes, cet aspect peut conduire à une mauvaise évaluation des performances. Par exemple, si notre algorithme de reconnaissance de fleur était entraîné sur un jeu de données comprenant des photos de tulipes annotées comme des photos d’iris (par exemple sur des clichés pris de loin), les performances du modèle s’en ressentiraient certainement.
Le syndrome de la boîte noire
Il est à ce jour impossible de connaître a priori l’architecture idéale d’un CNN* dédié à telle ou telle tâche. L’empirisme règne ici, extrêmement coûteux en temps de calcul (voir après ; Schwartz et al., 2019), chacun testant les performances de différents CNNs* et choisissant la meilleure option. Heureusement, les écologues se restreindront souvent à des architectures de modèles prédéfinies (par exemple ResNet-50), dans le cadre de l’apprentissage par transfert.
Par ailleurs, il est très difficile d’interpréter la capacité prédictive des CNNs* que l’on qualifie volontiers de boîtes noires (Voosen, 2017). Y aurait-il des critères compréhensibles par l’humain qui ont conduit à cette capacité ? Des zones des images particulièrement utiles (par exemple des éléments des ailes de papillons, Wu et al., 2019) ? Ou bien y aurait-il des éléments non souhaités et implicites contenus dans le contexte ou l’arrière-plan (Wearn, Freeman et Jacoby, 2019) qui rendraient le modèle moins généralisable ? Si, dans notre exemple, une pâquerette est prédite comme étant une tulipe, alors il n’y a pas d’élément facile à interroger pour comprendre les raisons de cette erreur. Il existe à ce jour quelques méthodes qui tentent d’interpréter les paramètres des modèles (Miao et al., 2019) mais ceci reste encore un sujet de recherche.
La technicité des codes de calcul
La communauté en apprentissage automatique, y compris issue des mastodontes du numériques, met à disposition de nombreux codes en libre accès qui sont réutilisables et modifiables. Ceci représente une excellente opportunité pour les écologues, sauf que ces codes sont écrits majoritairement dans le langage Python alors que les écologues plébiscitent le langage R. Sur une note optimiste, il faut signaler que l’apprentissage de Python est abordable, facilité par le grand nombre de ressources en ligne disponibles. Ensuite, le niveau de sophistication des algorithmes est tel que l’utilisation des bibliothèques logicielles du domaine (TensorFlow, Keras, PyTorch) est incontournable : un effort supplémentaire pour maîtriser ces bibliothèques logicielles est donc requis. Enfin, l’essor du deep learning* est lié à l’essor du calcul sur processeur graphique (GPU), matériel composé de milliers de petites unités de calcul, initialement dédié au jeu vidéo.
Les calculs matriciels associés à la convolution sont parfaitement adaptés aux contraintes des GPU et des gains de vitesse de l’ordre de x100 (dans nos expériences, de quelques minutes à quelques heures respectivement) sont à attendre entre une carte graphique et un ordinateur classique. Mais, ce matériel est difficile à installer et à gérer sans compétence informatique. Il est d’ailleurs rarement présent dans les laboratoires d’écologie. Matériel peu cher, il est très gourmand en électricité et l’installation d’un processeur graphique sur le poste informatique de chaque écologue serait anachronique dans un contexte de réduction des émissions de gaz à effet de serre. Nous recommandons ici de se tourner vers les différents centres académiques de mutualisation de moyens de calcul (par exemple, le calculateur CNRS national Jean Zay ou le centre régional GriCad).
Conclusions
Les techniques de deep learning* pour la vision par ordinateur suscitent un intérêt teinté d’une curiosité légitime. Nous appelons ici à une approche pragmatique visant à évaluer, en conscience, leurs forces et faiblesses au regard d’une utilisation en écologie. Nous rappelons tout de même que l’œil humain reste indispensable compte tenu des étapes d’annotation (en amont) et de validation (en aval) d’un modèle prédictif. L’utilisation massive de la machine serait aussi à coupler avec des discussions éthiques sur les potentiels dangers induits (Wearn, Freeman et Jacoby, 2019 ; Lamba et al., 2019). Par exemple, la diffusion d’information sur le positionnement de certaines espèces pourrait potentiellement être exploitée par des braconniers (Wearn, Freeman et Jacoby, 2019). Enfin, dans le cadre des programmes de production participative (crowdsourcing), le désir de s’inscrire dans une démarche pédagogique afin de sensibiliser les citoyens peut également conduire à ne pas se tourner vers la machine.
Nous nous risquons ici à quelques conseils pour des personnes désireuses de s’essayer au deep learning* :
0/ bien saisir le principe de l’apprentissage automatique* (pas nécessairement profond* ; voir l’exposé de Jennifer Hoeting) ;
1/ commencer par des cas simples et bien balisés, la plupart du temps pour automatiser une tâche facile mais fastidieuse avec la masse de données. Plusieurs besoins actuels peuvent être résolus, au moins partiellement (c’est-à-dire par exemple en prétraitement avant analyse manuelle), par des approches simples qui font des cas d’école pour les développeurs en deep learning* (voir le package R queyras, ou les tutoriels du site https://ecostat.gitlab.io/imaginecology/) ;
2/ apprendre les bases du langage Python en comprenant des petits codes disponibles (par exemple les cours de Joseph Salmon, http://josephsalmon.eu/HLMA408.html) ;
3/ promouvoir l’interdisciplinarité en constituant des groupes de travail avec différentes sensibilités scientifiques, depuis les écologues jusqu’à des spécialistes de l’analyse d’image et de l’informatique.
La maîtrise progressive des différents concepts et techniques par le plus grand nombre permettra une évaluation collective de ces approches et du bien-fondé de leur utilisation en écologie. C’est une démarche que nous initions actuellement au sein du GDR Ecologie Statistique et qui nous a conduits à rassembler quelques 200 collègues pour des premiers échanges sur le sujet (Novembre 2020, https://imaginecology.sciencesconf.org).
Glossaire :
Apprentissage automatique / machine learning : un algorithme est dit d’apprentissage automatique s’il a la capacité d’apprendre automatiquement des structures dans les données sans qu’elles soient nécessairement explicitées.
Apprentissage profond / deep learning : techniques d’apprentissage automatique* faisant usage de très nombreux niveaux de traitement du signal pour identifier des structures dans les données.
CNN : Convolutional Neural Networks, i.e. réseaux de neurones convolutifs en français. Modèle mathématique utilisé pour l’apprentissage profond. En particulier, il est basé sur la convolution qui consiste à utiliser une fenêtre glissante sur les données.
Modèle linéaire multinomial : modèle statistique liant la probabilité qu’un objet appartienne à une des catégories modélisées aux valeurs de variables explicatives associées aux objets présents dans les données, par exemple la probabilité qu’une image de tête de mésange bleue représente un mâle en fonction de la largeur du collier.
Problème de classification : problème consistant à assigner des données à des catégories (classes).
Spectrogramme : représentation des intensités et des fréquences d’un signal dans le temps, avec souvent le temps en abscisse, la fréquence en ordonnée et l’intensité sous forme de couleurs (voir Fig. 6), d’intensité de gris ou comme une troisième dimension.
Remerciements
Les auteurs remercient chaleureusement François Massol pour ses excellentes remarques et suggestions, ainsi qu’Anne Teyssèdre pour son aide à l’édition de ce ‘regard’. VM remercie le LECA-Chambéry pour l’avoir hébergé dans ses locaux. OG a bénéficié d’un soutien de la « Mission pour l’interdisciplinarité » du CNRS via son programme « Osez l’interdisciplinarité » et de l’ANR (ANR-16-CE02-0007).
Bibliographie
Abrams, J. F., A. Vashishtha et al., 2019. Habitat-Net: Segmentation of habitat images using deep learning. Ecological informatics 51, 121-128.
Anses, 2020. La saison de cueillette des champignons commence : restez vigilants face aux risques d’intoxications !
Ärje, J., C. Melvad et al., 2020. Automatic image-based identification and biomass estimation of invertebrates. Methods in Ecology and Evolution.
Baraniuk, R., D. Donoho & M. Gavish, 2020. The science of deep learning. Proceedings of the National Academy of Sciences 117, 30029-30032.
Beery, S., D. Morris & S. Yang, 2019. Efficient pipeline for camera trap image review. arXiv preprint arXiv:1907.06772.
Beery, S., G. Van Horn & P. Perona. Recognition in terra incognita. Proceedings of the European Conference on Computer Vision (ECCV), 456-473.
Bellman, R., 1966. Dynamic programming. Science 153, 34-37.
Bogucki, R., M. Cygan et al., 2019. Applying deep learning to right whale photo identification. Conservation Biology 33, 676-684.
Bolger, D. T., T. A. Morrison et al., 2012. A computer-assisted system for photographic mark–recapture analysis. Methods in Ecology and Evolution 3, 813-822.
Brodrick, P. G., A. B. Davies & G. P. Asner, 2019. Uncovering ecological patterns with convolutional neural networks. Trends in ecology and evolution 34, 734-745.
Charpentier, M. J. E., M. Harté et al., 2020. Same father, same face: Deep learning reveals selection for signaling kinship in a wild primate. Science Advances 6, eaba3274-eaba3274.
Christin, S., É. Hervet & N. Lecomte, 2019. Applications for deep learning in ecology. Methods in Ecology and Evolution 10, 1632-1644.
Duporge, I., O. Isupova et al., 2020. Using very-high-resolution satellite imagery and deep learning to detect and count African elephants in heterogeneous landscapes. Remote Sensing in Ecology and Conservation.
Dutta, A. & A. Zisserman, 2019. The VIA annotation software for images, audio and video Proceedings of the 27th ACM International Conference on Multimedia:2276-2279.
Ferreira, A. C., L. R. Silva et al., 2020. Deep learning-based methods for individual recognition in small birds. Methods in Ecology and Evolution.
Graving, J. M., D. Chae et al., 2019. DeepPoseKit, a software toolkit for fast and robust animal pose estimation using deep learning. eLife 8, e47994-e47994.
Guirado, E., S. Tabik et al., 2019. Whale counting in satellite and aerial images with deep learning. Scientific Reports 9, 1-12.
Huang, H., H. Zhou, X. Yang, L. Zhang, L. Qi & A.-Y. Zang, 2019. Faster R-CNN for marine organisms detection and recognition using data augmentation. Neurocomputing 337, 372-384.
Jin, H., Q. Song & X. Hu. Auto-keras: An efficient neural architecture search system. Proceedings of the 25th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining,1946-1956.
Kattenborn, T., J. Eichel & F. E. Fassnacht, 2019. Convolutional Neural Networks enable efficient, accurate and fine-grained segmentation of plant species and communities from high-resolution UAV imagery. Scientific Reports 9, 1-9.
Konieczny, S. & H. Prade, 2020. L’intelligence Artificielle : De quoi s’agit-il vraiment ?, Cépaduès.
Krizhevsky, A., I. Sutskever & G. E. Hinton. Imagenet classification with deep convolutional neural networks Advances in neural information processing systems:1097-1105.
Lamba, A., P. Cassey, R. R. Segaran & L. P. Koh, 2019. Deep learning for environmental conservation. Current Biology 29, R977-R982.
Mac Aodha, O., R. Gibb et al., 2018. Bat detective—Deep learning tools for bat acoustic signal detection. PLoS Computational Biology 14, e1005995-e1005995.
Masteling, R., L. Voorhoeve et al., 2020. DiSCount: computer vision for automated quantification of Striga seed germination. Plant Methods 16, 1-8.
Mathis, A., P. Mamidanna et al., 2018. DeepLabCut: markerless pose estimation of user-defined body parts with deep learning. Nature Neuroscience 21, 1281-1289.
Metz, C., 2019. AI is learning from humans. Many humans. The New York Times 16. https://www.nytimes.com/2019/08/16/technology/ai-humans.html.
Miao, Z., K. M. Gaynor et al., 2019. Insights and approaches using deep learning to classify wildlife. Scientific Reports 9, 1-9.
Miele, V., G. Dussert et al., 2021. Revisiting giraffe photo-identification using deep learning and network analysis. Methods in Ecology and Evolution.
Norouzzadeh, M. S., A. Nguyen et al., 2018. Automatically identifying, counting, and describing wild animals in camera-trap images with deep learning. Proceedings of the National Academy of Sciences 115, E5716-E5725.
O’Neil, C. 2016, Weapons of math destruction: How big data increases inequality and threatens democracy, Broadway Books.
Pereira, T. D., D. E. Aldarondo et al., 2019. Fast animal pose estimation using deep neural networks. Nature Methods 16, 117-125.
Schneider, S., S. Greenberg, G. W. Taylor & S. C. Kremer, 2020. Three critical factors affecting automated image species recognition performance for camera traps. Ecology and Evolution 10, 3503-3517.
Schneider, S., G. W. Taylor & S. Kremer. Deep learning object detection methods for ecological camera trap data 2018 15th Conference on computer and robot vision (CRV):321-328.
Schneider, S., G. W. Taylor, S. Linquist & S. C. Kremer, 2019. Past, present and future approaches using computer vision for animal re-identification from camera trap data. Methods in Ecology and Evolution 10, 461-470.
Schofield, D., A. Nagrani et al., 2019. Chimpanzee face recognition from videos in the wild using deep learning. Science Advances 5, eaaw0736-eaaw0736.
Schwartz, R., J. Dodge, N. A. Smith & O. Etzioni, 2019. Green AI. arXiv preprint arXiv:1907.10597.
Sethi, S. S., N. S. Jones et al., 2020. Characterizing soundscapes across diverse ecosystems using a universal acoustic feature set. Proceedings of the National Academy of Sciences 117, 17049-17055.
Stowell, D., M. D. Wood et al., 2019. Automatic acoustic detection of birds through deep learning: the first Bird Audio Detection challenge. Methods in Ecology and Evolution 10, 368-380.
Suresh, H. & J. V. Guttag, 2019. A framework for understanding unintended consequences of machine learning. arXiv preprint arXiv:1901.10002.
Torney, C. J., D. J. Lloyd-Jones et al., 2019. A comparison of deep learning and citizen science techniques for counting wildlife in aerial survey images. Methods in Ecology and Evolution 10, 779-787.
Villon, S., D. Mouillot et al., 2018. A deep learning method for accurate and fast identification of coral reef fishes in underwater images. Ecological informatics 48, 238-244.
Voosen, P., 2017. How AI detectives are craking open the black box of deep learning. Science.
Wearn, O. R., R. Freeman & D. M. P. Jacoby, 2019. Responsible AI for conservation. Nature Machine Intelligence 1, 72-73.
Willi, M., R. T. Pitman et al., 2019. Identifying animal species in camera trap images using deep learning and citizen science. Methods in Ecology and Evolution 10, 80-91.
Wu, S., C.-M. Chang et al., 2019. Artificial intelligence reveals environmental constraints on colour diversity in insects. Nature Communications 10, 1-9.
Voir aussi :
Detection d’animaux en temps réel dans une vidéo
L’IA par le journal du CNRS
Introduction aux réseaux de neurones profonds, par Stéphane Mallat
Tutoriel en 3 parties sur le deep learning pour l’image, par Vincent Miele
A Statistical View of Deep Learning in Ecology, par Jennifer Hoeting
Parallèle entre R et Python, très pratique pour apprendre Python
Calculateur d’émission de carbone avec une GPU
Regards connexes :
Regards sur les méthodes et outils : https://sfecologie.org/tag/methodes-et-outils/
—–
Regard édité par François Massol et Anne Teyssèdre.
Article publié également sur HAL, la plateforme de diffusion d’articles scientifiques du CNRS : https://hal.archives-ouvertes.fr/hal-03142486
——-
Forum de discussion sur ce regard

Commentaires récents